Parceria GIA-ATGC
No dia 09 de fevereiro de 2021 foi finalmente assinado o Termo de Compartilhamento estabelecido entre a UFPR, através de...
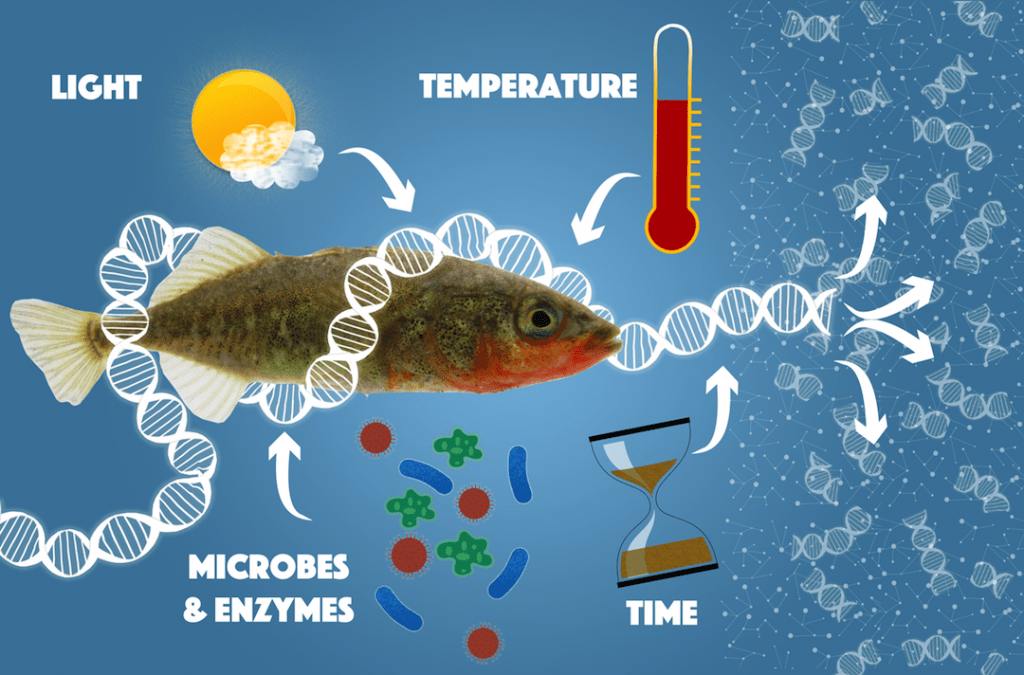
A biologia encontra-se em meio de uma verdadeira revolução. O sequenciamento de DNA, outrora uma técnica cara e demorada, tem se tornado cada vez mais rápido, sensível, e capaz de produzir quantidades cada vez maiores de dados genéticos. Por exemplo, um único exemplar de equipamento de alta performance de sequenciamento de DNA (Illumina HiSeq 250) possui a capacidade de produção de dados equivalente a todos os centros de pesquisa no mundo em 2006, sendo atualmente capaz de gerar 1 trilhão de bases em uma única corrida. Estes avanços têm possibilitado oportunidades sem precedentes na detecção e monitoramento de espécies em ambientes silvestres.
Um destes avanços envolve o estudo de DNA ambiental (environmental DNA ou eDNA), que está fundamentado na detecção de moléculas de DNA produzidas como subproduto das atividades de uma determinada espécie, como aquelas encontradas em fezes, muco, saliva ou outra fonte de células mortas. Métodos de laboratório podem ser empregados para detectar e quantificar estas moléculas, levando a diversos tipos de aplicações. Um dos casos mais extensivos do uso de eDNA envolve o US Army Corps of Engineers . Através desta iniciativa, duas espécies de peixes invasores (Hypophthalmichthys nobilis e H. molitrix) tem sido monitoradas. Tendo em vista os potenciais efeitos negativos destas espécies sobre recursos pesqueiros e ambientes naturais da região dos Grandes Lagos e bacias hidrográficas adjacentes o monitoramento destas espécies tem se tornado uma ferramenta indispensável para medidas eficientes de manejo e controle das espécies invasoras.
Neste caso, é necessário primeiro adaptar, testar e validar o método para espécies de peixes de água doce, especialmente em ambientes tropicais de alta diversidade, como é o caso dos rios brasileiros, pois somente após a validação da efetividade do método é que ele poderá ser usado em substituição à metodologia tradicional de monitoramento ictiofaunístico.
Integrar os conhecimentos já gerados sobre a ictiofauna do reservatório de Itaipu e do Canal da Piracema com avanços recentes na tecnologia de sequenciamento de DNA ambiental, a partir do desenvolvimento de um método preciso e inovador de monitoramento da ictiofauna local.

 Recursos naturais na vida caiçara (livro digital - PDF)
R$0,00
Recursos naturais na vida caiçara (livro digital - PDF)
R$0,00
 Aquicultura no Brasil: o desafio é crescer (Livro Digital -PDF)
R$0,00
Aquicultura no Brasil: o desafio é crescer (Livro Digital -PDF)
R$0,00
 Piscicultura - Fundamentos e Técnicas de Manejo (Livro Digital - PDF)
R$0,00
Piscicultura - Fundamentos e Técnicas de Manejo (Livro Digital - PDF)
R$0,00
 Cartilha de Rastreabilidade: Ostras cultivadas - Projeto AquiNordeste (Livro Digital - PDF).
Cartilha de Rastreabilidade: Ostras cultivadas - Projeto AquiNordeste (Livro Digital - PDF).
 Aquicultura: Uma visão geral sobre a produção de organismos aquáticos no Brasil... (Livro digital - PDF)
R$0,00
Aquicultura: Uma visão geral sobre a produção de organismos aquáticos no Brasil... (Livro digital - PDF)
R$0,00
 A produção integrada na carcinicultura brasileira (Volume 2) - (Livro Digital - PDF)
R$0,00
A produção integrada na carcinicultura brasileira (Volume 2) - (Livro Digital - PDF)
R$0,00
 A produção integrada na carcinicultura brasileira (Volume 1) - (Livro Digital - PDF)
R$0,00
A produção integrada na carcinicultura brasileira (Volume 1) - (Livro Digital - PDF)
R$0,00
 Manual de ostreicultura com espécies nativas da região Nordeste do Brasil (livro digital - PDF)
R$0,00
Manual de ostreicultura com espécies nativas da região Nordeste do Brasil (livro digital - PDF)
R$0,00
 Manual de Boas Práticas: Qualidade e Segurança para Bons Negócios (livro digital - PDF)
R$0,00
Manual de Boas Práticas: Qualidade e Segurança para Bons Negócios (livro digital - PDF)
R$0,00
 Fichas Técnicas Ilustradas - Sebrae/Projeto AquiNordeste (Livro Digital - PDF)
R$0,00
Fichas Técnicas Ilustradas - Sebrae/Projeto AquiNordeste (Livro Digital - PDF)
R$0,00
O GIA é um grupo de pesquisas científicas, tecnológicas e de inovação criado e sediado na Universidade Federal do Paraná. Sua principal missão é assumir um papel direto na interação entre setor acadêmico, a iniciativa privada e a sociedade civil em geral, transpondo os muros universitários, aplicando com excelência e eficiência, métodos técnico-científicos de produção de alimentos e na resolução de problemas ambientais, sociais e econômicos que dizem respeito aos mais diferentes setores da sociedade brasileira.
No dia 09 de fevereiro de 2021 foi finalmente assinado o Termo de Compartilhamento estabelecido entre a UFPR, através de...
O GIA acaba de lançar mais um livro. O tema da vez é o cultivo de peixes orgânicos Cada vez...
Copyright © Grupo Integrado de Aquicultura e Estudos Ambientais (GIA) | Desenvolvido por Abrigo Virtual
Copyright © Grupo Integrado de Aquicultura e Estudos Ambientais (GIA) | Desenvolvido por Abrigo Virtual