Parceria GIA-ATGC
No dia 09 de fevereiro de 2021 foi finalmente assinado o Termo de Compartilhamento estabelecido entre a UFPR, através de...
Publicado em 30/08/2018
Por Aline Horodesky
As espécies exóticas invasoras são organismos não-nativos que habitam uma determinada área, ameaçam negativamente os ecossistemas, habitats e outras espécies, sendo hoje, consideradas uma das maiores ameaças da biodiversidade do planeta(9).

Figura 1: Esquema mostrando a invasão do mexilhão-zebra no ecossistema. Fonte: Cary Institute of Ecosystem Studies
O estudo de bioinvasão concentra-se, em geral, em avaliações dos impactos que as espécies invasoras ocasionam sobre os ecossistemas locais ou diretamente sobre a gestão dessas espécies invasoras, o que inclui a previsão de quais espécies são propensas a se tornar invasoras, buscando impedir a introdução de tais espécies e tentando controlar ou conviver com as invasões incipientes. Sua ciência é complexa e multidisciplinar e requer a consideração de muitos processos, incluindo aspectos comportamentais, ecológicos, genéticos e econômicos e pode ser dividida em três grandes áreas: prevenção, predição e controle(8).
Como a gestão ambiental ou operacional ligada a espécies invasoras pode envolver elevados custos financeiros e econômicos, é essencial que as estratégias escolhidas facilitem a avaliação rápida e precisa dos indivíduos e permita a tomada de decisões mais efetivas e mais eficientes em relação à identificação do potencial invasivo das espécies, a avaliação do impacto das invasões, ao estabelecimento de mecanismos que possam impedir as invasões e ao controle das espécies invasoras que tenham sido recentemente introduzidas(5).
Os métodos convencionais de detecção de organismos invasores são baseados na sua identificação visual, o que é pouco eficiente, demorado e caro(12). Além disso, poucas são as ferramentas que auxiliam na amostragem de animais com baixa abundância em um determinado ambiente(2). O exemplo mais evidente é o fato de se ainda utilizar métodos visuais (microscopia óptica) para a identificação de invertebrados aquáticos. Este método, além de muito lento e impreciso, exige gastos com equipes qualificadas de coleta, fixação de amostras e identificação dos organismos coletados.
Atualmente, novos métodos vêm sendo testados e validados para a utilização com este propósito: detecção de espécies únicas por PCR tradicional e PCR quantitativa (qPCR); detecção de múltiplas espécies por metabarcoding (Sequenciamento de Nova Geração-NGS). Em ambos os métodos, é necessária a utilização do ácido desoxirribonucleico (DNA). Esse DNA é liberado pelos organismos em ambientes aquáticos ou terrestres, a partir de renovações celulares, pela urina, fezes e descamação epidérmica(11;6). Após sua liberação ele passa a ser chamado de DNA ambiental ou eDNA(10).
Quando liberado no ambiente esse DNA pode ser degradado por DNAses microbioanas e utilizado como nutriente para plantas, pode ser incorporado a um genoma microbiano como fonte de informação ou pode persistir no ambiente. A utilização do eDNA, mesmo sem quaisquer sinais evidentes de material de origem biológica é um eficiente, não-invasivo e facilmente padronizável meio de amostragem.
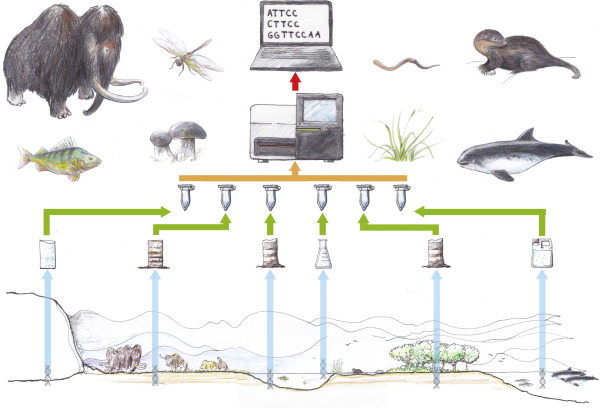
Figura 2: Esquema de utilização de DNA ambiental (eDNA) para detecção de espécies. Fonte: Thomsen & Willerslev, 2015.
O DNA obtido diretamente de amostras ambientais foi aplicado primeiramente para avaliar sedimentos antigos, revelando o passado de mamíferos, aves e plantas extintos(15). Atualmente, esse eDNA é utilizado na análise de sedimentos terrestres, núcleos de gelo, lagos e rios de água doce(4;3), principalmente com o objetivo de conservação do ecossistema.
Para as espécies exóticas invasoras, esse novo método está sendo utilizado para monitor as taxas de invasão de carpas asiáticas (Hypophthalmichthys molitrix, Hypophthalmichthys nobilis), para o monitoramento de espécies de peixes(7), anuros Lithobates catesbeianus(1) e invertebrados, como o lagostim Procambarus clarkii(14) e o molusco Potamopyrcus antipodarum(2).
Desta forma, com essas novas ferramentas, substituem-se as análises visuais e morfológicas, por análises genéticas moleculares, elevando-se a eficiência na identificação e quantificação das espécies-alvo com uma precisão que só é possível se obter através do método molecular e, ainda sim, reduzindo o tempo e os custos das análises.
Referências consultadas
1. Dejean, T.; Valentini, A.; Miquel, C.; Taberlet, P.; Bellemain, E.; Miaud, C. 2012. Improved detection of an alien invasive species through environmental DNA barcoding: the example of the American bullfrog Lithobates catesbeianus. Journal of Applied Ecology, 49, 953-959.
2. Goldberg, C. S.; Sepulveda, A.; Ray, A.; Baumgardt, J.; Waits, L. P. 2013. Environmental DNA as a new method for early detection of New Zealand mudsnails (Potamopyrgus antipodarum). Freshwater Science, 32(3):792-800.
3. Haile, J.; Holdaway, R.; Oliver, K.; Bunce, M.; Gilbert, M. T. P.; Nielsen, R.; Munch, K.; Ho, S. Y. W.; Shapiro, B.; Willerslev, E. 2007. Ancient DNA chronology within sediment deposits: are paleobiological reconstructions possible and is DNA leaching a factor? Molecular Biology and Evolution, 24, 982-989.
4. Halpern, B. S. 2008. A Global Map of Human Impact on Marine Ecosystems. Science 319, 5865:948-952.
5. Hofreiter, M.; Mead, J. I.; Martin, P.; Poinar, H. N. 2003. Molecular caving. Current Biology, 13, 693-695.
6. Jerde, C. L.; Mahon, A. R.; Chadderton, W. L.; Lodge, D. M. 2011. “Sight‐unseen” detection of rare aquatic species using environmental DNA. Conservation Letters, 4, 150-157.
7. Lydolph, M. C.; Jacobsen, J.; Arctander, P.; Gilbert, M. T. P.; Gilichinsky, D. A.; Hansen, A. J.; Willerslev, E.; Lange, L. 2005. Beringian paleoecology inferred from permafrost-preserved fungal DNA. Applied and Environmental Microbiology, 71, 1012-1017.
8. Mahon, A. R.; Jerde, C. L.; Galaska, M.; Bergner, J. L.; Chadderton, W. L.; Lodge, D. M.; Hunter, M. E.; Nico, L. G. 2013. Validation of eDNA surveillance sensitivity for detection of asian carps in controlled and field experiments. PLoS ONE 8, 58316.
9. McCormick, F. H.; Contreras, G. C.; Johnson, S. L. 2009. Effects of Nonindigenous Invasive Species on Water Quality and Quantity. A Dynamic Invasive Species Research Vision: Opportunities and Priorities, 29, p. 111-120.
10. Ministério do Meio Ambiente, Secretaria de Biodiversidade e Florestas. 2006. Espécies exóticas invasoras: Situação brasileira. Brasília.
11. Nielsen, K. M.; Johnsen, P. J.; Bensasson, D.; Daffonchio, D. 2007. Release and persistence of extracellular DNA in the environment. Environmental Biosafety Research, 6, 37-53.
12. Poinar, H. N.; Hofreiter, M.; Spaulding, W. G.; Martin, P. S.; Stankiewicz, B. A.; Bland, H.; Evershed, R. P.; Possnert, G.; Pääbo, S. 1998. Molecular coproscopy: dung and diet of the extinct ground sloth nothrotheriops shastensis. Science, 281, 402-406.
13. Thomsen, P. F.; Kielgast, J.; Iversen, L. L.; Wiuf, C.; Rasmussen, M.; Gilbert, M. T. P.; Orlando, L.; Willerslev, E. 2012. Monitoring endangered freshwater biodiversity using environmental DNA. Molecular Ecology, 21, 2565-2573.
14. Thomsen, P. F.; Willerslev, E. 2015. Environmental DNA – An emerging tool in conservation for monitoring past and present biodiversity. Biological Conservation 183, 4-18.
15. Tréguier, A.; Paillisson, J. M.; Dejean, T.; Valentini, A.; Schlaepfer, M. A.; Roussel, J. M. 2014. Environmental DNA surveillance for invertebrate species: advantages and technical limitations to detect invasive crayfish Procambarus clarkii in freshwater ponds. Journal of Applied Ecology, 51, 871-879.
16. Willerslev, E.; Hansen, A. J.; Binladen, J.; Brand, T. B.; Gilbert, M. T. P.; Shapiro, B.; Bunce, M.; Wiuf, C.; Gilichinsky, D. A.; Cooper, A. 2003. Diverse plant and animal genetic records from holocene and pleistocene sediments. Science, 300, 791-795.
 Cartilha de Rastreabilidade: Ostras cultivadas - Projeto AquiNordeste (Livro Digital - PDF).
Cartilha de Rastreabilidade: Ostras cultivadas - Projeto AquiNordeste (Livro Digital - PDF).
 A produção integrada na carcinicultura brasileira (Volume 2) - (Livro Digital - PDF)
R$0,00
A produção integrada na carcinicultura brasileira (Volume 2) - (Livro Digital - PDF)
R$0,00
 As lendas na educação - Estórias do Baixo Sul e do Recôncavo Baiano (livro digital - PDF)
R$0,00
As lendas na educação - Estórias do Baixo Sul e do Recôncavo Baiano (livro digital - PDF)
R$0,00
 Manual de Boas Práticas: Qualidade e Segurança para Bons Negócios (livro digital - PDF)
R$0,00
Manual de Boas Práticas: Qualidade e Segurança para Bons Negócios (livro digital - PDF)
R$0,00
 Piscicultura - Fundamentos e Técnicas de Manejo (Livro Digital - PDF)
R$0,00
Piscicultura - Fundamentos e Técnicas de Manejo (Livro Digital - PDF)
R$0,00
 Água no setor industrial da região do Alto Iguaçu e dos afluentes do Alto Ribeira... (Versão Digital - PDF)
R$0,00
Água no setor industrial da região do Alto Iguaçu e dos afluentes do Alto Ribeira... (Versão Digital - PDF)
R$0,00
 A produção integrada na carcinicultura brasileira (Volume 1) - (Livro Digital - PDF)
R$0,00
A produção integrada na carcinicultura brasileira (Volume 1) - (Livro Digital - PDF)
R$0,00
 Manual de ostreicultura com espécies nativas da região Nordeste do Brasil (livro digital - PDF)
R$0,00
Manual de ostreicultura com espécies nativas da região Nordeste do Brasil (livro digital - PDF)
R$0,00
 FAO: Agricultura Irrigada Sustentável no Brasil - Identificação de Áreas... (Livro Digital - PDF)
R$0,00
FAO: Agricultura Irrigada Sustentável no Brasil - Identificação de Áreas... (Livro Digital - PDF)
R$0,00
 As lendas na educação caiçara (livro digital - PDF)
R$0,00
As lendas na educação caiçara (livro digital - PDF)
R$0,00
O GIA é um grupo de pesquisas científicas, tecnológicas e de inovação criado e sediado na Universidade Federal do Paraná. Sua principal missão é assumir um papel direto na interação entre setor acadêmico, a iniciativa privada e a sociedade civil em geral, transpondo os muros universitários, aplicando com excelência e eficiência, métodos técnico-científicos de produção de alimentos e na resolução de problemas ambientais, sociais e econômicos que dizem respeito aos mais diferentes setores da sociedade brasileira.
No dia 09 de fevereiro de 2021 foi finalmente assinado o Termo de Compartilhamento estabelecido entre a UFPR, através de...
O GIA acaba de lançar mais um livro. O tema da vez é o cultivo de peixes orgânicos Cada vez...
Copyright © Grupo Integrado de Aquicultura e Estudos Ambientais (GIA) | Desenvolvido por Abrigo Virtual
Copyright © Grupo Integrado de Aquicultura e Estudos Ambientais (GIA) | Desenvolvido por Abrigo Virtual