Parceria GIA-ATGC
No dia 09 de fevereiro de 2021 foi finalmente assinado o Termo de Compartilhamento estabelecido entre a UFPR, através de...
Por Ana Paula da Silva Bertão
O e-DNA ou DNA ambiental é uma mistura complexa de DNA genômico oriundo de organismos inteiros ou partes deles, presentes em amostras ambientais. Essas amostras podem ser, por exemplo, de solo, água ou sedimento. O e-DNA permite a caracterização e o monitoramento da composição de espécies em ambientes de difícil coleta, como em rios, mares e baías. Sua principal aplicação é a análise ambiental, pois permite obter informações genômicas de diversas espécies.
A técnica de e-DNA foi desenvolvida em 1987 para a detecção de DNA microbiano em sedimento. Contudo, essa técnica passou a ser mais utilizada a partir do século XXI, quando os primeiros sequenciadores de DNA começaram a ser comercializados em meados de 2005.
Com os avanços da genética aplicada associadas à eficiência dos levantamentos de dados em e-DNA, essa ferramenta tem se tornado peça fundamental para a conservação de espécies nativas e para o monitoramento de ecossistemas aquáticos.
Para a identificação dos táxons presentes em uma amostra com e-DNA, é utilizada a técnica da Reação em Cadeia da Polimerase (PCR). A PCR tem a finalidade de produzir uma grande quantidade de cópias de uma determinada parte do DNA, ou seja, consiste na amplificação “in vitro” de uma região específica de DNA.
Este método ocorre em três etapas:
Desnaturação- quando acontece a abertura das duas fitas do DNA (esta etapa ocorre com temperatura a 95º C).
Anelamento ou ligação dos primers- os primes ou iniciadores se ligam em uma das extremidades de cada fita separada, para a enzima polimerase iniciar a síntese da nova fita (60 a 64º C).
Extensão ou elongação- acontece a síntese da nova fita realizada pela enzima polimerase (72º C). Toda esta metodologia ocorre em aproximadamente 40 a 60 ciclos promovendo a amplificação da região que se pretende avaliar, conforme ilustrado na Figura 1.
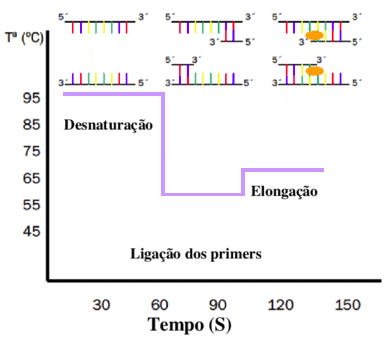
Figura 1-Etapas da Reação em Cadeia da Polimerase PCR (Sanchéz, 2012).
Para a quantificação dos táxons utiliza-se a PCR em Tempo Real ou também denominada qPCR. A qPCR é uma variante da reação de PCR convencional, em que a amplificação e a detecção dos pares de bases dos nucleotídeos ocorrem simultaneamente. Consequentemente, o resultado é visualizado em tempo real durante a amplificação da sequência de interesse, com a possibilidade de gerar resultados quantitativos com maior precisão. Essa quantificação é mensurada pela quantidade de produtos amplificados durante cada ciclo (pela fluorescência emitida). Esta técnica utiliza um equipamento com sistema de monitoramento da emissão de fluorescência, como por exemplo o Termociclador de PCR em Tempo Real (qPCR).
A qPCR ou amplificação ocorre em três etapas:
Crescimento exponencial: acontece de forma rápida e precisa, devido à especificidade que a sonda fluorescente possui.
Crescimento linear: os produtos da reação são consumidos e iniciam o processo de degradação.
Estacionária: corresponde ao final da análise devido ao elevado nível de degradação dos produtos da PCR, ilustrada na Figura 2.
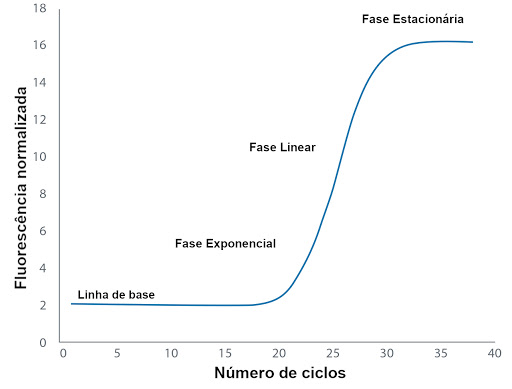
Figura 2- Etapas da amplificação a partir da qPCR (Arya et al. 2005).
O SYBR Green, é um corante do ácido desoxirribonucleico (DNA), utilizado na técnica de qPCR, que é adicionado ao DNA, gerando um sinal de fluorescência. Essa fluorescência é diretamente proporcional à quantidade de produto amplificado ao longo das diferentes fases.
As análises ambientais que utilizam a qPCR, possuem algumas vantagens que refletem nos resultados finais da análise. Pode-se citar o acompanhamento da reação em tempo real, em que os resultados são mais rápidos e precisos, quando comparados a outros métodos convencionais, permite a detecção de mais de um DNA alvo, facilita a quantificação e diminui a contaminação das amostras ambientais.
Aplicações do e-DNA
Pode ser aplicado em diversas áreas da pesquisa e tem sido fortemente recomendado para a identificação de espécies invasoras aquáticas, principalmente as de difícil detecção em fases iniciais do ciclo de vida. A detecção precoce destas espécies é considerada crucial para o manejo e controle da bioinvasão.
No Brasil, são encontradas duas espécies invasoras incrustantes oriundas do continente asiático, o molusco invasor mexilhão-dourado Limnoperna fortunei e o hidrozoário de água doce Cordylophora caspia. Ambas espécies possuem comportamento invasivo, incrustante e depositam material biológico para se instalarem/fixarem nos ambientes invadidos, causando diversos impactos econômicos e ecológicos.
Os impactos econômicos estão associados aos danos causados pelo entupimento de trocadores de calor de usinas hidrelétricas, estações de captação de água, bombas e tubulações. Os impactos ecológicos, são direcionados as espécies aquáticas nativas, que são afetadas e prejudicadas pela concorrência por espaços e alimentos, mudanças na estrutura da cadeia trófica e perturbação das comunidades aquáticas.
A detecção destas espécies através da técnica de e-DNA tem possibilitado ótimos resultados quanto à identificação, avaliação, monitoramento e proteção da biodiversidade local, permitindo melhor gerenciamento e conservação dos ecossistemas aquáticos. As vantagens das análises com e-DNA são evidenciadas quando comparadas aos métodos convencionais de identificação de organismos, como a utilização de lupas e microscópio estereoscópio. Pois, os métodos convencionais envolvem resultados morosos e propensos a erros durante a seleção de larvas ou na identificação das espécies.
Referências
ARYA, M. et al. Basic principles of real-time quantitative PCR. Expert review of molecular diagnostics, v. 5, n. 2, p. 209-219, 2005. ISSN 1473-7159.
DARRIGRAN, G.; DAMBORENEA, C. Introdução a biologia das invasões o Mexilhão Dourado na América do Sul: biologia, dispersão, impacto, prevenção e controlo. CUBO Editora, São Carlos, 2009.
DONIA, D. T. qRT-PCR for enterovirus detection: Conversion to ultrafast protocols. Journal of King Saud University-Science, v. 30, n. 2, p. 180-184, 2018. ISSN 1018-3647.
FERNANDES, F. D. et al. Abordagem conceitual dos moluscos invasores nos ecossistemas límnicos brasileiros. Porto Alegre: Redes Editora, 2012. 412 ISBN 978-85-61638-46-7.
LATINI, A. et al. Espécies exóticas invasoras de águas continentais no Brasil. Brasília: MMA, 2016.
MANSUR, M. C. D. et al. Moluscos límnicos – bivalves. 2016. ISBN 978-85-7738-176-0.
NASCIMENTO, S.; SUAREZ, E. R.; PINHAL, M. A. D. S. Tecnologia de PCR e RT
-PCR em tempo real e suas aplicações na área médica. Revista Brasileira de Medicina, v. 67, p. 7-19, 2010.
OGRAM, A.; SAYLER, G. S.; BARKAY, T. The extraction and purification of microbial DNA from sediments. Journal of microbiological methods, v. 7, n. 2-3, p. 57-66, 1987. ISSN 0167-7012.
PESTANA, D. et al. Prospecção do molusco invasor Limnoperna fortunei (Dunker, 1857) nos principais corpos hídricos do estado do Paraná, Brasil. Papéis Avulsos de Zoologia (São Paulo), v. 50, n. 34, p. 553-559, 2010. ISSN 0031-1049.
PIE, M. R. et al. Development of a real-time PCR assay for the detection of the golden mussel (Limnoperna fortunei, Mytilidae) in environmental samples. Anais da Academia Brasileira de Ciências, v. 89, n. 2, p. 1041-1045, 2017. ISSN 0001-3765.
REES, H. C. et al. The detection of aquatic animal species using environmental DNA–a review of eDNA as a survey tool in ecology. Journal of Applied Ecology, v. 51, n. 5, p. 1450-1459, 2014. ISSN 1365-2664.
SÁNCHEZ, A. C. Identificación y cuantificación de especies del género Merluccius mediante la utilización de PCR a tiempo real. 2012.
TABERLET, P. et al. Environmental DNA: For Biodiversity Research and Monitoring. Oxford University Press, 2018. ISBN 0191079995.
THOMSEN, P. F.; WILLERSLEV, E. Environmental DNA–An emerging tool in conservation for monitoring past and present biodiversity. Biological Conservation, v. 183, p. 4-18, 2015. ISSN 0006-3207.
 Recursos naturais na vida caiçara (livro digital - PDF)
R$0,00
Recursos naturais na vida caiçara (livro digital - PDF)
R$0,00
 A produção integrada na carcinicultura brasileira (Volume 2) - (Livro Digital - PDF)
R$0,00
A produção integrada na carcinicultura brasileira (Volume 2) - (Livro Digital - PDF)
R$0,00
 As lendas na educação - Estórias do Baixo Sul e do Recôncavo Baiano (livro digital - PDF)
R$0,00
As lendas na educação - Estórias do Baixo Sul e do Recôncavo Baiano (livro digital - PDF)
R$0,00
 As lendas na educação caiçara (livro digital - PDF)
R$0,00
As lendas na educação caiçara (livro digital - PDF)
R$0,00
 Atlas Anatômico e Histológico do Caranguejo-uçá (Livro Impresso)
Atlas Anatômico e Histológico do Caranguejo-uçá (Livro Impresso)
 Cartilha de Rastreabilidade: Ostras cultivadas - Projeto AquiNordeste (Livro Digital - PDF).
Cartilha de Rastreabilidade: Ostras cultivadas - Projeto AquiNordeste (Livro Digital - PDF).
 Água no setor industrial da região do Alto Iguaçu e dos afluentes do Alto Ribeira... (Versão Digital - PDF)
R$0,00
Água no setor industrial da região do Alto Iguaçu e dos afluentes do Alto Ribeira... (Versão Digital - PDF)
R$0,00
 Piscicultura - Fundamentos e Técnicas de Manejo (Livro Digital - PDF)
R$0,00
Piscicultura - Fundamentos e Técnicas de Manejo (Livro Digital - PDF)
R$0,00
 Rastreabilidade na ostreicultura: conceitos, fundamentos e casos... (livro digital - PDF)
R$0,00
Rastreabilidade na ostreicultura: conceitos, fundamentos e casos... (livro digital - PDF)
R$0,00
 Manual de ostreicultura com espécies nativas da região Nordeste do Brasil (livro digital - PDF)
R$0,00
Manual de ostreicultura com espécies nativas da região Nordeste do Brasil (livro digital - PDF)
R$0,00
O GIA é um grupo de pesquisas científicas, tecnológicas e de inovação criado e sediado na Universidade Federal do Paraná. Sua principal missão é assumir um papel direto na interação entre setor acadêmico, a iniciativa privada e a sociedade civil em geral, transpondo os muros universitários, aplicando com excelência e eficiência, métodos técnico-científicos de produção de alimentos e na resolução de problemas ambientais, sociais e econômicos que dizem respeito aos mais diferentes setores da sociedade brasileira.
No dia 09 de fevereiro de 2021 foi finalmente assinado o Termo de Compartilhamento estabelecido entre a UFPR, através de...
O GIA acaba de lançar mais um livro. O tema da vez é o cultivo de peixes orgânicos Cada vez...
Copyright © Grupo Integrado de Aquicultura e Estudos Ambientais (GIA) | Desenvolvido por Abrigo Virtual
Copyright © Grupo Integrado de Aquicultura e Estudos Ambientais (GIA) | Desenvolvido por Abrigo Virtual