Parceria GIA-ATGC
No dia 09 de fevereiro de 2021 foi finalmente assinado o Termo de Compartilhamento estabelecido entre a UFPR, através de...

Por: Aline Horodesky
O estudo de DNA ambiental (eDNA), está baseado na detecção de moléculas de DNA produzidas como subproduto das atividades de uma determinada espécie, como aquelas encontradas em fezes, muco, saliva ou outra fonte de células mortas. Métodos de laboratório podem ser empregados para detectar e quantificar estas moléculas, levando a diversos tipos de aplicações. Uma potencial extensão da abordagem de eDNA envolve a detecção de mais de uma espécie simultaneamente.
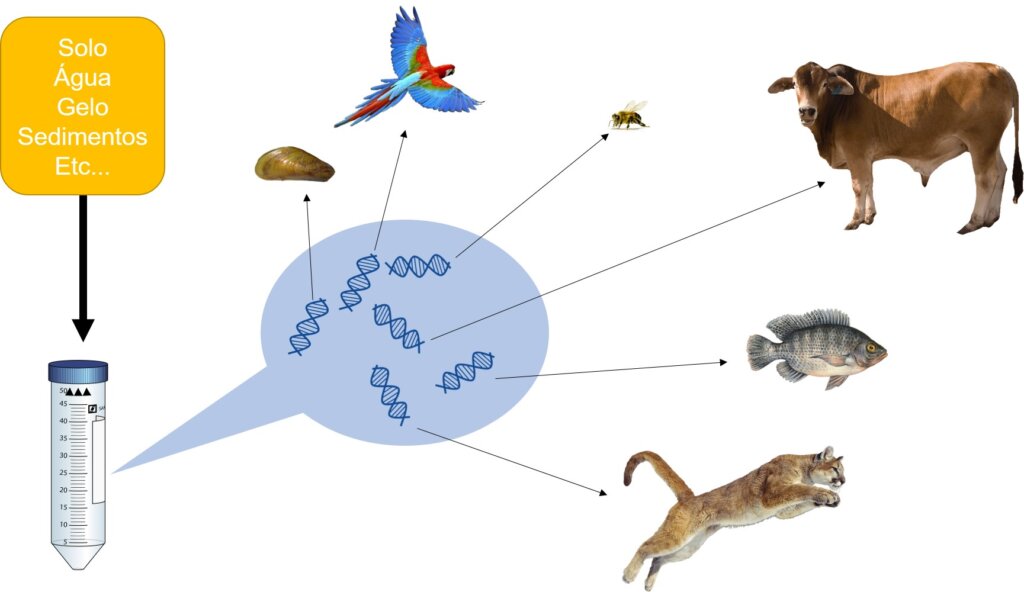
Figura 1. Esquema mostrando a detecção de diferentes espécies a partir de uma amostra ambiental.
A primeira referência ao DNA ambiental remonta a 1987 e diz respeito a um método para extração de DNA microbiano de sedimentos. No entanto, o termo realmente surgiu no início dos anos 2000, principalmente usado pela comunidade de microbiologistas. DNA ambiental refere-se ao DNA que pode ser extraído de amostras ambientais (como solo, água ou ar), sem primeiro isolar qualquer organismo-alvo. É caracterizado por uma mistura complexa de DNA genômico de muitos organismos diferentes e por possível degradação (isto é, por moléculas de DNA que são cortadas em pequenos fragmentos). O eDNA total contém DNA celular originado de células ou organismos vivos e DNA extracelular resultante da morte celular natural e subsequente destruição da estrutura celular.
Microbiologistas têm analisado o eDNA do solo ou da água há mais de uma década, o que lhes deu acesso à genética de microrganismos não cultiváveis. Eles tinham três objetivos principais: (i) identificar táxons microbianos presentes em amostras ambientais; (ii) identificar as funções bioquímicas mais importantes por meio da análise de genes codificadores; e (iii) montar genomas inteiros de microrganismos não cultiváveis. A análise do eDNA por microbiologistas foi denominada metagenômica, que apenas se referia à montagem e análises funcionais (objetivos ii e iii) do eDNA de dados de sequências, mas não identificações baseadas na amplificação e sequenciação de genes marcadores como o 16S rDNA (objetivo i).
Contudo, a utilização do eDNA passou a se disseminar principalmente a partir de 2011, com aprimoramentos constantes significativos a cada ano. A identificação de táxons baseada em DNA foi estendida para a invertebrados da meiofauna (por exemplo, nematóides) e para microrganismos, usando uma abordagem de PCR/clonagem ou PCR/sequenciamento de segunda geração, isso já no início dos anos 2000, mas com o único objetivo de identificar os diferentes organismos que contribuíam com o DNA de uma determinada amostra.
Nos últimos anos, avanços significativos estão sendo alcançados nesta área de pesquisa. A coleta de amostras de eDNA realizadas em parques de safári, jardins zoológicos e fazendas, demonstraram que análises do DNA do solo podem ser usadas para registrar a presença de grandes mamíferos. Inventários de minhocas feitos através de métodos tradicionais consomem muito tempo e são muito complexos. Mas, uma tecnologia desenvolvida nos Alpes franceses e usando eDNA já permite que se trabalhe com comunidades de minhocas em qualquer parte do mundo.
Claramente, o surgimento de eDNA em estudos ecológicos mais amplos está hoje ligado à disponibilidade de sequenciadores de próxima geração (NGS), que permitem contornar a etapa cara e demorada de clonagem e sequenciamento de produtos de PCR usando o sequenciamento Sanger. Não há dúvidas de que, a partir de agora, as análises de eDNA serão integradas a mais e mais estudos ecológicos. O sequenciamento de DNA, outrora uma técnica cara e demorada, tem se tornado cada vez mais rápido, sensível, e capaz de produzir quantidades cada vez maiores de dados genéticos. Por exemplo, um único equipamento de alta performance de sequenciamento de DNA (Illumina HiSeq 250) possui hoje a mesma capacidade de produção de dados que possuíam conjuntamente todos os centros de pesquisa no mundo em 2006, sendo atualmente capaz de gerar 1 trilhão de bases em uma única corrida. Estes avanços têm possibilitado oportunidades sem precedentes na detecção e monitoramento de espécies em ambientes silvestres.
Uma potencial extensão da abordagem de eDNA envolve a detecção de mais de uma espécie simultaneamente. No futuro, espera-se que as abordagens baseadas em eDNA possam ser utilizadas como marcador único para análises de espécies ou de comunidades, através de pesquisas de metagenômica, de ecossistemas inteiros, possibilitando a prevenção dos padrões de biodiversidade espaciais e temporais.
Referências consultadas
YU, D. W. et al. Biodiversity soup: metabarcoding of arthropods for rapid biodiversity assessment and biomonitoring. Methods in Ecology and Evolution, v. 3, n. 4, p. 613-623, 2012.
FOOTE, A. D. et al. Investigating the Potential Use of Environmental DNA (eDNA) for Genetic Monitoring of Marine Mammals. PLOS ONE, v. 7, n. 8, p. e41781, 2012.
MINAMOTO, T. et al. Surveillance of fish species composition using environmental DNA. Limnology, v. 13, n. 2, p. 193-197, August 01 2012. ISSN 1439-863X.
MÄCHLER, E. et al. Utility of environmental DNA for monitoring rare and indicator macroinvertebrate species. Freshwater Science, v. 33, n. 4, p. 1174-1183, 2014.
NOAA Ocean Exploration. Environmental DNA (eDNA). ThayerMahan, Inc., Kraken Robotics and NOAA Office of Ocean Exploration and Research, 2021.
KELLY, R. P. et al. Using Environmental DNA to Census Marine Fishes in a Large Mesocosm. PLOS ONE, v. 9, n. 1, p. e86175, 2014.
TABERLET, P. et al. Environmental DNA. Molecular Ecology, v. 21, n. 8, p. 1789-1793, 2012/04/01 2012. ISSN 0962-1083.
BRADY, S. F. Construction of soil environmental DNA cosmid libraries and screening for clones that produce biologically active small molecules. Nature protocols, v. 2, n. 5, p. 1297, 2007.
ROBE, P. et al. Extraction of DNA from soil. European Journal of Soil Biology, v. 39, n. 4, p. 183-190, 2003.
DOPHEIDE, A. et al. Impacts of DNA extraction and PCR on DNA metabarcoding estimates of soil biodiversity. Methods in Ecology and Evolution, v. 10, n. 1, p. 120-133, 2019.
DEJEAN, T. et al. Persistence of Environmental DNA in Freshwater Ecosystems. PLOS ONE, v. 6, n. 8, p. e23398, 2011
NICHOLS, R. V. et al. Browsed twig environmental DNA: diagnostic PCR to identify ungulate species. Molecular Ecology Resources, v. 12, n. 6, p. 983-989, 2012.
BARNES, M. A. et al. Environmental Conditions Influence eDNA Persistence in Aquatic Systems. Environmental Science & Technology, v. 48, n. 3, p. 1819-1827, 2014/02/04 2014.
DEINER, K.; ALTERMATT, F. Transport Distance of Invertebrate Environmental DNA in a Natural River. PLOS ONE, v. 9, n. 2, p. e88786, 2014.
PILLIOD, D. S. et al. Factors influencing detection of eDNA from a stream-dwelling amphibian. Molecular Ecology Resources, v. 14, n. 1, p. 109-116, 2014.
WILCOX, T. M. et al. A blocking primer increases specificity in environmental DNA detection of bull trout (Salvelinus confluentus). Conservation Genetics Resources, v. 6, n. 2, p. 283-284, 2014.
BLAXTER, M. L. The promise of a DNA taxonomy. Philosophical Transactions of the Royal Society of London. Series B: Biological Sciences, v. 359, n. 1444, p. 669-679, 2004.
BLAXTER, M. Molecular systematics: counting angels with DNA. Nature, v. 421, n. 6919, p. 122, 2003.
BLAXTER, M. et al. Defining operational taxonomic units using DNA barcode data. Philosophical Transactions of the Royal Society B: Biological Sciences, v. 360, n. 1462, p. 1935-1943, 2005.
USHIO, M. et al. Environmental DNA enables detection of terrestrial mammals from forest pond water. Molecular Ecology Resources, v. 17, n. 6, p. e63-e75, 2017.
NICHOLS, R. V. et al. Browsed twig environmental DNA: diagnostic PCR to identify ungulate species. Molecular Ecology Resources, v. 12, n. 6, p. 983-989, 2012
BOESSENKOOL, S. et al. Blocking human contaminant DNA during PCR allows amplification of rare mammal species from sedimentary ancient DNA. Molecular ecology, v. 21, n. 8, p. 1806-1815, 2012.
BIENERT, F. et al. Tracking earthworm communities from soil DNA. Molecular Ecology, v. 21, n. 8, p. 2017-2030, 2012.
KITSON, J. J. et al. Detecting host–parasitoid interactions in an invasive Lepidopteran using nested tagging DNA metabarcoding. Molecular ecology, v. 28, n. 2, p. 471-483, 2019.
RAGUPATHY, S. et al. Exploring DNA quantity and quality from raw materials to botanical extracts. Heliyon, v. 5, n. 6, p. e01935, 2019.
DARLING, J. A.; MAHON, A. R. From molecules to management: Adopting DNA-based methods for monitoring biological invasions in aquatic environments. v. 111, n. 7, p. 978-988, 2011.
THOMSEN, P. F. et al. Detection of a Diverse Marine Fish Fauna Using Environmental DNA from Seawater Samples. PLOS ONE, v. 7, n. 8, p. e41732, 2012.
YOCCOZ, N. G. The future of environmental DNA in ecology. Molecular Ecology, v. 21, n. 8, p. 2031-2038, 2012.
WILCOX, T. M. et al. Robust Detection of Rare Species Using Environmental DNA: The Importance of Primer Specificity. PLOS ONE, v. 8, n. 3, p. e59520, 2013.
TAKAHARA, T. et al. Estimation of Fish Biomass Using Environmental DNA. PLOS ONE, v. 7, n. 4, p. e35868, 2012.
 Camarões Marinhos - Engorda: Roberto Carlos Barbieri Jr. & Antonio Ostrensky (Livro Impresso)
R$55,00
Camarões Marinhos - Engorda: Roberto Carlos Barbieri Jr. & Antonio Ostrensky (Livro Impresso)
R$55,00
 Aquicultura: Uma visão geral sobre a produção de organismos aquáticos no Brasil... (Livro digital - PDF)
R$0,00
Aquicultura: Uma visão geral sobre a produção de organismos aquáticos no Brasil... (Livro digital - PDF)
R$0,00
 Camarões Marinhos Reprodução, Maturação e Larvicultura (Livro Impresso)
R$50,00
Camarões Marinhos Reprodução, Maturação e Larvicultura (Livro Impresso)
R$50,00
 FAO: Agricultura Irrigada Sustentável no Brasil - Identificação de Áreas... (Livro Digital - PDF)
R$0,00
FAO: Agricultura Irrigada Sustentável no Brasil - Identificação de Áreas... (Livro Digital - PDF)
R$0,00
 Rastreabilidade na ostreicultura: conceitos, fundamentos e casos... (livro digital - PDF)
R$0,00
Rastreabilidade na ostreicultura: conceitos, fundamentos e casos... (livro digital - PDF)
R$0,00
 A produção integrada na carcinicultura brasileira (Volume 1) - (Livro Digital - PDF)
R$0,00
A produção integrada na carcinicultura brasileira (Volume 1) - (Livro Digital - PDF)
R$0,00
 Aquicultura no Brasil: o desafio é crescer (Livro Digital -PDF)
R$0,00
Aquicultura no Brasil: o desafio é crescer (Livro Digital -PDF)
R$0,00
 Piscicultura - Fundamentos e Técnicas de Manejo (Livro Digital - PDF)
R$0,00
Piscicultura - Fundamentos e Técnicas de Manejo (Livro Digital - PDF)
R$0,00
 Manual de ostreicultura com espécies nativas da região Nordeste do Brasil (livro digital - PDF)
R$0,00
Manual de ostreicultura com espécies nativas da região Nordeste do Brasil (livro digital - PDF)
R$0,00
 As lendas na educação - Estórias do Baixo Sul e do Recôncavo Baiano (livro digital - PDF)
R$0,00
As lendas na educação - Estórias do Baixo Sul e do Recôncavo Baiano (livro digital - PDF)
R$0,00
O GIA é um grupo de pesquisas científicas, tecnológicas e de inovação criado e sediado na Universidade Federal do Paraná. Sua principal missão é assumir um papel direto na interação entre setor acadêmico, a iniciativa privada e a sociedade civil em geral, transpondo os muros universitários, aplicando com excelência e eficiência, métodos técnico-científicos de produção de alimentos e na resolução de problemas ambientais, sociais e econômicos que dizem respeito aos mais diferentes setores da sociedade brasileira.
No dia 09 de fevereiro de 2021 foi finalmente assinado o Termo de Compartilhamento estabelecido entre a UFPR, através de...
O GIA acaba de lançar mais um livro. O tema da vez é o cultivo de peixes orgânicos Cada vez...
Copyright © Grupo Integrado de Aquicultura e Estudos Ambientais (GIA) | Desenvolvido por Abrigo Virtual
Copyright © Grupo Integrado de Aquicultura e Estudos Ambientais (GIA) | Desenvolvido por Abrigo Virtual