Parceria GIA-ATGC
No dia 09 de fevereiro de 2021 foi finalmente assinado o Termo de Compartilhamento estabelecido entre a UFPR, através de...
Por: Camila Duarte Ritter
Já imaginou poder identificar uma comunidade inteira de peixes em um rio sem a necessidade de pescá-los? Atualmente isso é possível utilizando o DNA ambiental ou eDNA (do inglês environmental DNA). Esse eDNA consiste no DNA deixado por organismos através de fezes, muco, gametas, peles, carcaças, pelos, entre outros, em uma variedade de amostras ambientais, como solo, água ou até mesmo no ar1. Assim, ao coletar amostras ambientais, como a água de um rio, podemos analisá-las por métodos de sequenciamento de DNA de alto rendimento, como, por exemplo, o metabarcoding2, possibilitando o monitoramento e medição rápidos da biodiversidade.
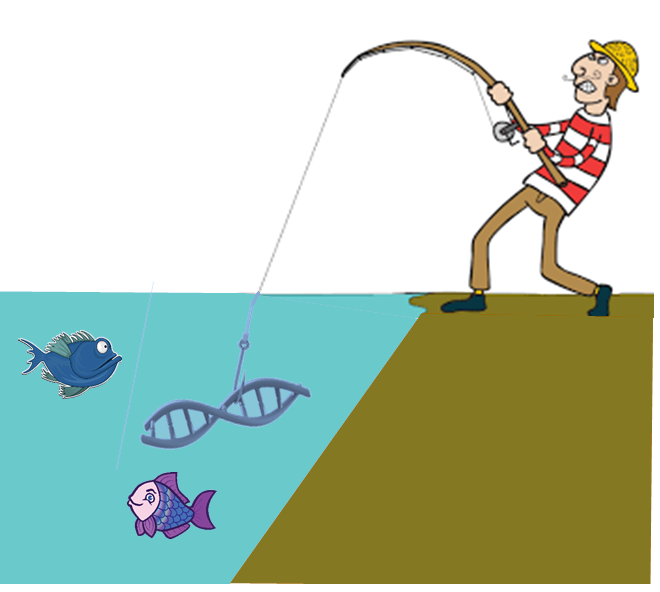
Figura 1. Todos os organismos deixam traços de DNA no ambiente no qual ocorrem. Esse DNA pode ser “pescado” e sequenciado para se determinar a presença dessas espécies.
Para amostrar espécies com o metabarcoding, primeiramente as amostras são coletadas, por exemplo um litro de água de um rio, após o DNA é extraído e, em seguida, amplificado usando primers que determinam o fragmento de DNA alvo na reação em cadeia da polimerase e, por fim, sequenciado usando sequenciamento de próxima geração para gerar milhares a milhões de leituras desse DNA. Para identificar as espécies em uma amostra, as sequências de DNA registradas são comparadas a bibliotecas de DNA previamente estudadas e depositadas em banco de referência, como o GenBank3. A partir desses dados, a presença de espécies pode ser determinada e a biodiversidade geral avaliada.
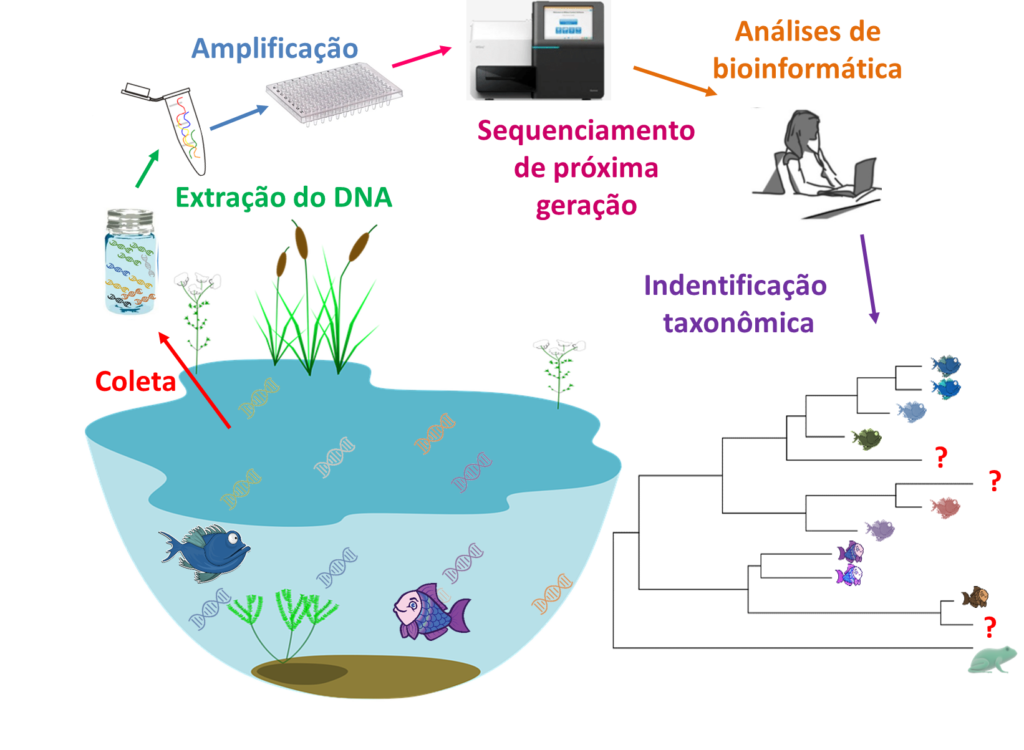
Figura 2. Processo de identificação de espécies usando DNA ambiental e metabarcoding. Para identificar as espécies é necessário se ter sequências disponíveis, caso contrario as sequências registradas não serão identificadas.
A análise de eDNA tem grande potencial, não apenas para monitorar espécies comuns, mas para detectar geneticamente e identificar outras espécies existentes, como raras, ameaçadas ou invasoras, que podem influenciar os esforços de conservação4. Este método permite o biomonitoramento sem exigir a coleta do organismo vivo, facilitando a amostragem de uma forma não invasiva (sem necessidade de manipulação ou abate). É importante também ressaltar que o metabarcoding de eDNA costuma ser mais econômico em comparação aos métodos tradicionais de amostragem5.
A Usina Hidrelétrica de Itaipu, construída na fronteira do Brasil com o Paraguai, é a segunda maior do mundo em geração de energia6, perdendo apenas para a Usina das Três Gargantas na China. Com a formação e enchimento de seu reservatório, em 1982, a barreira natural para a migração de peixes do trecho médio do rio Paraná (queda de Sete Quedas) foi substituída pela barreira artificial da barragem de Itaipu. Essa barreira artificial causou impactos nas assembléias de peixes adjacentes, como a redução da atividade reprodutiva nos primeiros quilômetros a jusante da barragem7. Para permitir a migração de peixes e mitigar o impacto ambiental da barragem, um sistema de passagem de peixes conhecido como Canal da Piracema foi criado em 2002, ligando o Rio Paraná ao Reservatório de Itaipu8. Para para monitorar o impacto da barragem e do Canal da Piracema nas comunidades de peixes do rio Paraná, utilizamos DNA ambiental de amostras de água, coletadas em 2019 e 2020, e metabarcoding e comparamos com os métodos tradicionais de monitoramento.
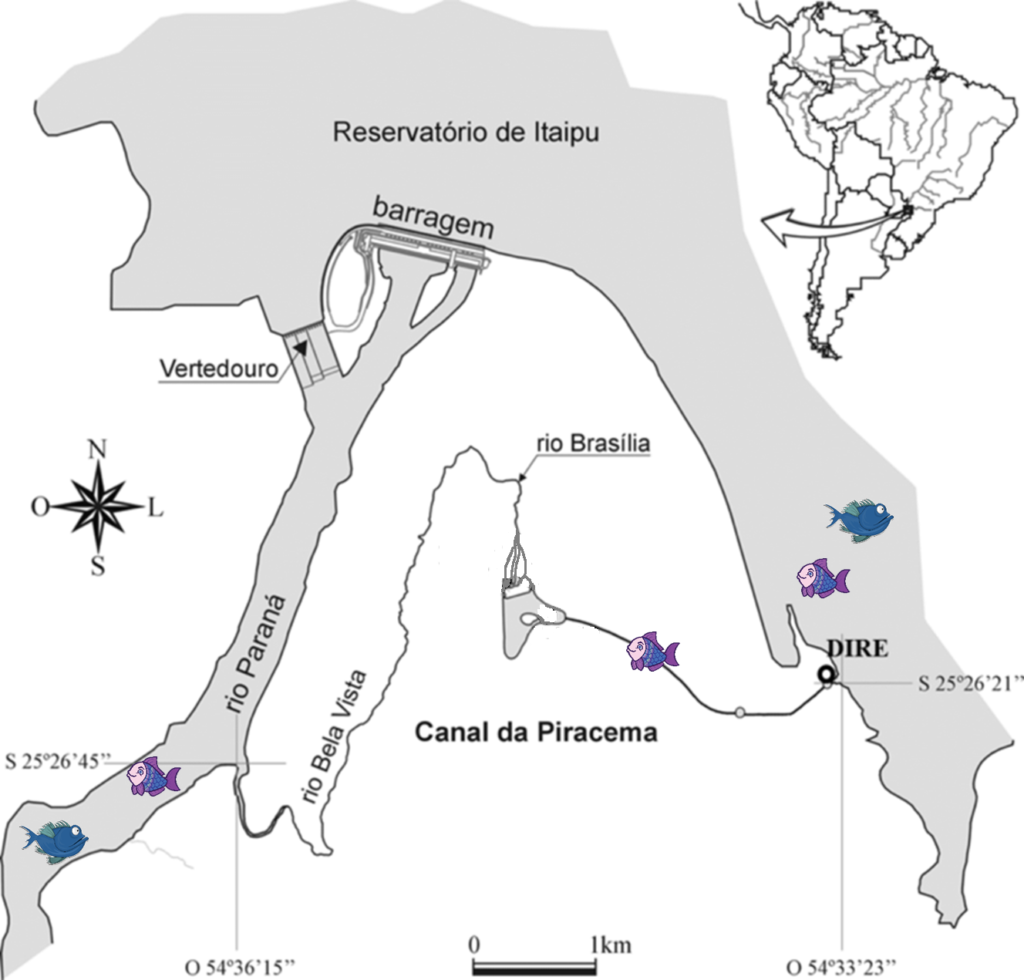
Figura 3. Canal da Piracema conectando o Rio Paraná ao Reservatório de Itaipu.
Usando um fragmento do gene mitocondrial 12S, caracterizamos a riqueza de espécies e a composição da comunidade de peixes com base nas sequências geradas por metabarcoding. Com esse método registramos 123 espécies do qual 37 foram identificadas com base em sequências disponíveis. Amostragem tradicional realizada em cinco anos, integrando quatro métodos amostrais, registrou 136 espécies de peixes. Ou seja, em duas ocasiões amostrais rápidas registram os 90% das espécies registradas historicamente no canal. Também encontramos uma diminuição na diversidade de peixes de 2019 a 2020, com os dados de metabarcoding, provavelmente devido à recente seca extrema experimentada no sudeste do Brasil. A maior diversidade de espécies foi registrada na boca do canal, no Rio Paraná, localizado em um vale protegido com a maior heterogenedade ambiental. Apesar da indicação clara de que os bancos de dados de referência precisam ser continuamente melhorados, nossos resultados demonstram a eficiência analítica do metabarcoding para monitorar espécies de peixes.
Quer saber mais sobre esse estudo? Ele está disponivel aqui:
Dal Pont, G., Ritter, C. D., Agostinis, A. O., Stika, P. V., Horodesky, A., Cozer, N., … & Pie, M. R. (2021). Monitoring fish communities through DNA metabarcoding in the fish pass system of the second largest hydropower plant in the world. bioRxiv.
https://www.biorxiv.org/content/10.1101/2021.08.17.456687v1.full.pdf
Referências
 Água na indústria: uma análise da visão do setor industrial... (Versão Digital - PDF)
R$0,00
Água na indústria: uma análise da visão do setor industrial... (Versão Digital - PDF)
R$0,00
 Água no setor industrial da região do Alto Iguaçu e dos afluentes do Alto Ribeira... (Versão Digital - PDF)
R$0,00
Água no setor industrial da região do Alto Iguaçu e dos afluentes do Alto Ribeira... (Versão Digital - PDF)
R$0,00
 Manual de ostreicultura com espécies nativas da região Nordeste do Brasil (livro digital - PDF)
R$0,00
Manual de ostreicultura com espécies nativas da região Nordeste do Brasil (livro digital - PDF)
R$0,00
 FAO: Agricultura Irrigada Sustentável no Brasil - Identificação de Áreas... (Livro Digital - PDF)
R$0,00
FAO: Agricultura Irrigada Sustentável no Brasil - Identificação de Áreas... (Livro Digital - PDF)
R$0,00
 Manual de Boas Práticas: Qualidade e Segurança para Bons Negócios (livro digital - PDF)
R$0,00
Manual de Boas Práticas: Qualidade e Segurança para Bons Negócios (livro digital - PDF)
R$0,00
 Um pouco de tudo o que você sempre quis saber sobre o cultivo de peixes orgânicos (livro digital - PDF)
R$0,00
Um pouco de tudo o que você sempre quis saber sobre o cultivo de peixes orgânicos (livro digital - PDF)
R$0,00
 Rastreabilidade na ostreicultura: conceitos, fundamentos e casos... (livro digital - PDF)
R$0,00
Rastreabilidade na ostreicultura: conceitos, fundamentos e casos... (livro digital - PDF)
R$0,00
 Aquicultura: Uma visão geral sobre a produção de organismos aquáticos no Brasil... (Livro digital - PDF)
R$0,00
Aquicultura: Uma visão geral sobre a produção de organismos aquáticos no Brasil... (Livro digital - PDF)
R$0,00
 Piscicultura - Fundamentos e Técnicas de Manejo (Livro Digital - PDF)
R$0,00
Piscicultura - Fundamentos e Técnicas de Manejo (Livro Digital - PDF)
R$0,00
 Camarões Marinhos Reprodução, Maturação e Larvicultura (Livro Impresso)
R$50,00
Camarões Marinhos Reprodução, Maturação e Larvicultura (Livro Impresso)
R$50,00
O GIA é um grupo de pesquisas científicas, tecnológicas e de inovação criado e sediado na Universidade Federal do Paraná. Sua principal missão é assumir um papel direto na interação entre setor acadêmico, a iniciativa privada e a sociedade civil em geral, transpondo os muros universitários, aplicando com excelência e eficiência, métodos técnico-científicos de produção de alimentos e na resolução de problemas ambientais, sociais e econômicos que dizem respeito aos mais diferentes setores da sociedade brasileira.
No dia 09 de fevereiro de 2021 foi finalmente assinado o Termo de Compartilhamento estabelecido entre a UFPR, através de...
O GIA acaba de lançar mais um livro. O tema da vez é o cultivo de peixes orgânicos Cada vez...
Copyright © Grupo Integrado de Aquicultura e Estudos Ambientais (GIA) | Desenvolvido por Abrigo Virtual
Copyright © Grupo Integrado de Aquicultura e Estudos Ambientais (GIA) | Desenvolvido por Abrigo Virtual