Parceria GIA-ATGC
No dia 09 de fevereiro de 2021 foi finalmente assinado o Termo de Compartilhamento estabelecido entre a UFPR, através de...
Por: Camila Ritter
Existem atualmente 1,5 milhão de espécies de eucariotos descritas e cerca de 11 milhões estimadas na Terra, embora a estimativa possa variar entre 2 milhões a 1 trilhão (Mora et al. 2011, Tedersoo et al. 2021). Por mais impressionantes que sejam esses números, eles são ofuscados pela riqueza estimada de procariontes, estimada entre 106-108 espécies (Ward 2002). Há um forte viés em nossa compreensão da biodiversidade: devido à facilidade de amostragem e identificação em alguns grupos taxonômicos, quase toda a sua diversidade esperada foi formalmente descrita, como aves – cerca de 98% das espécies e mamíferos – cerca de 99% (Chapman 2009). No entanto, a esmagadora maioria da biodiversidade existente não pertence a esses grupos. Todos os vertebrados combinados representam apenas 0,7% das espécies estimadas de eucariotos, enquanto 23-34% são representados por insetos (Hamilton et al. 2010) e 14-28% por fungos (Fig. 1; Tedersoo et al. 2021).
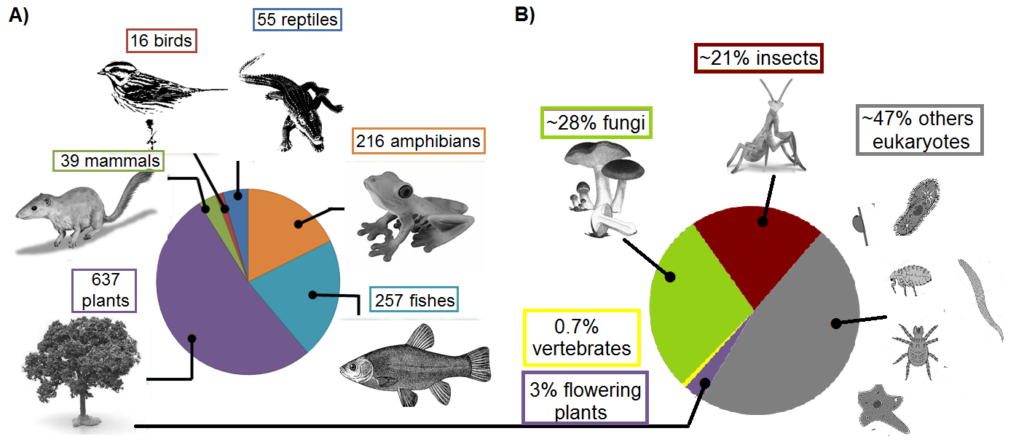
Figura 1. Comparação do número de espécies recentemente descritas na Amazônia e a proporção de plantas com flores e vertebrados em relação a todos os eucariotos descritos. A) Número de novas espécies descritas na Amazônia entre 1999 e 2009 de acordo com um relatório do WWF (2013). B) Proporção aproximada dos grupos de eucariontes mais bem estudados em relação a todos os eucariontes. A região em amarelo representa todos os vertebrados combinados (0,7% de todos os eucariotos). A fatia cinza indica os outros eucariotos (por exemplo, protistas, nematóides, moluscos).
Os dados moleculares revolucionaram o estudo científico de grupos de organismos imperceptíveis e, atualmente, os dados de sequência de DNA são uma importante fonte de informação sobre a evolução e ecologia de microrganismos (Creer et al. 2016, Doliwa et al. 2020). As avaliações da biodiversidade usando métodos moleculares formam uma ferramenta poderosa para entender comunidades inteiras e começar a entender suas interações bióticas/abióticas. No entanto, mais esforço e desenvolvimento de métodos são necessários para corrigir erros em bancos de dados de sequências de referência e as diferenças intrínsecas entre avaliações taxonômicas e moleculares da biodiversidade.
Há muitos fatores a serem considerados ao usar ferramentas moleculares em avaliações de biodiversidade, incluindo extração de DNA, escolha do marcador genético (Elbrecht et al. 2016), método de sequenciamento (Ritter et al. 2020) e procedimentos de análise de dados (Prodan et al. 2020). Uma séria ressalva para o uso desses métodos moleculares para avaliações de biodiversidade é a falta de bancos de dados de referência taxonômicos ricamente povoados, especialmente para as regiões tropicais do globo (Dal Pont et al. 2021). Sem esses bancos de dados de referência, as sequências recuperadas não podem ser combinadas com os níveis taxonômicos resolvidos. Além disso, a avaliação molecular é dificultada por sequências de referência anotadas incorretamente, sequências tecnicamente comprometidas (por exemplo, quimeras) e sequências de referência anotadas apenas em altos níveis taxonômicos (por exemplo filo; Nilsson et al. 2012). Algumas iniciativas recentes tentam mitigar esses problemas, incluindo, entre outros, os Observatórios Genômicos Globais (Davies et al. 2012) e bancos de dados para identificação molecular (Yilmaz et al. 2014, Abarenkov et al. 2016). Mas mesmo com esses esforços continua sendo um desafio avaliar corretamente muitas sequências recém-geradas.
A dificuldade em avaliar a biodiversidade é amplificada em ambientes megadiversos e mal amostrados, como florestas tropicais (Ritter et al. 2019a). Na floresta amazônica, por exemplo, centenas de novas espécies são descritas a cada ano (Maretti 2014, Barrett et al. 2018), e a taxa de degradação desse bioma é alarmantemente alta (Fig. 2; Pereira et al. 2019), provavelmente levando muitas espécies à extinção antes mesmo de serem descobertas e formalmente descritas (Wearn et al. 2012). Porém, mesmo em ambientes altamente diversificados e pouco amostrados para os quais os bancos de dados de referência são muito pouco povoados, o uso de unidades taxonômicas operacionais moleculares (OTUs; Blaxter et al. 2005) permite uma avaliação da diversidade genética e permite a comparação entre vários locais (Ritter et al. 2019b, c).
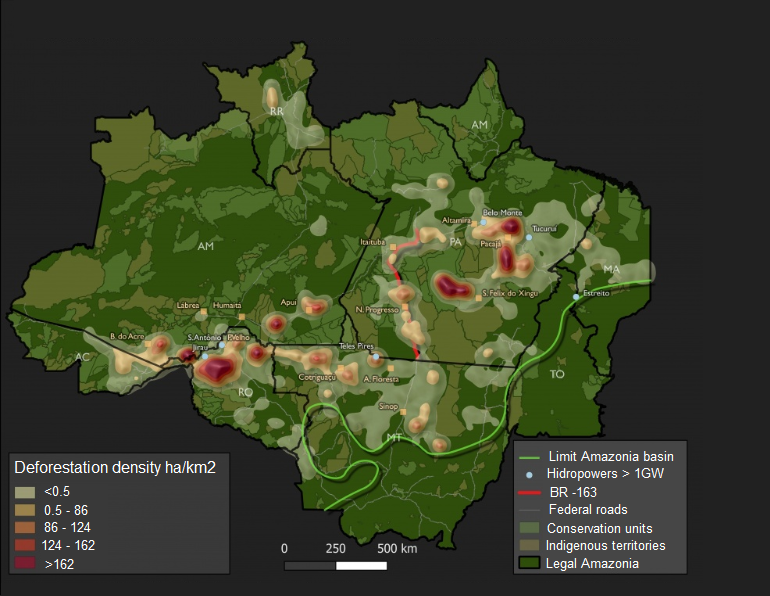
Figura 2. Densidade de desmatamento na Amazônia brasileira durante 2015 – 2016. Mapa modificado do Instituto Socioambiental – ISA com dados gerados a partir de: desmatamento do INPE/PRODES (2016), unidades de conservação e territórios indígenas do ISA (2016), estradas e Amazônia limites do IBGE (2012) e hidrelétricas da ANAEL (2016). Os dados foram coletados entre agosto de 2015 e julho de 2016 e mostraram quase 8 mil quilômetros quadrados de desmatamento, um aumento de cerca de 30% em relação ao ano anterior.
É importante destacar que, em geral, os dados moleculares derivados do “sequenciamento ambiental” devem ser vistos como complementares, ao invés de competir com os estudos taxonômicos. De fato, uma maior fusão de ambas as linhas de evidência é altamente justificada, pois será necessário superar suas respectivas deficiências (Dal Pont et al. 2021). Atualmente, partes da comunidade taxonômica dão pouca atenção (positiva) aos estudos de sequenciamento ambiental e vice-versa. É particularmente importante que os resultados da amostragem ambiental estejam disponíveis para os taxonomistas, pois podem incentivar o trabalho com linhagens não tratadas em uma estrutura taxonômica. Essa interação provavelmente criará “ciclos de retroalimentação taxonômica” que acelerará a descoberta de espécies e linhagens e potencialmente adicionará galhos proeminentes à árvore da vida. Assim, tal abordagem integrativa também melhorará a eficiência das estratégias de conservação, uma vez que mais esforços podem ser direcionados para “verdadeiros” hotspots abrangentes de biodiversidade, incluindo riqueza taxonômica e linhagens únicas em grupos “ocultos” combinando ambas abordagens taxonômica e moleculares de DNA ambiental.
Referencias:
Abarenkov, K. et al. 2016. Annotating public fungal ITS sequences from the built environment according to the MIxS-Built Environment standard – A report from a May 23-24, 2016 workshop (Gothenburg, Sweden). – MycoKeys in press.
Barrett, M. et al. 2018. Living planet report 2018: Aiming higher. in press.
Blaxter, M. et al. 2005. Defining operational taxonomic units using DNA barcode data.: 1935–1943.
Chapman, A. D. 2009. Numbers of living species in Australia and the world. in press.
Creer, S. et al. 2016. The ecologist’s field guide to sequence-based identification of biodiversity. – Methods Ecol. Evol. 7: 1008–1018.
Dal Pont, G. et al. 2021. Monitoring fish communities through environmental DNA metabarcoding in the fish pass system of the second largest hydropower plant in the world. – Sci. Rep. 11: 1–13.
Davies, N. et al. 2012. A call for an international network of genomic observatories (GOs). – Gigascience 1: 1–5.
Doliwa, A. et al. 2020. Identifying potential hosts of short-branch Microsporidia. – Microb. Ecol.: 1–5.
Elbrecht, V. et al. 2016. Testing the potential of a ribosomal 16S marker for DNA metabarcoding of insects. – PeerJ 4: e1966.
Hamilton, A. J. et al. 2010. Quantifying uncertainty in estimation of tropical arthropod species richness. – Am. Nat. 176: 90–95.
Maretti, C. C. 2014. Amazon: There is Hope! If we all do ‘the right thing’…; Deforestation, Protected Areas and Indigenous Territories: Past, evolution and… Which future?
Mora, C. et al. 2011. How many species are there on earth and in the ocean? – PLoS Biol. 9: 1–8.
Nilsson, R. H. et al. 2012. Five simple guidelines for establishing basic authenticity and reliability of newly generated fungal ITS sequences. – MycoKeys 4: 37–63.
Pereira, E. J. de A. L. et al. 2019. Policy in Brazil (2016–2019) threaten conservation of the Amazon rainforest. – Environ. Sci. Policy 100: 8–12.
Prodan, A. et al. 2020. Comparing bioinformatic pipelines for microbial 16S rRNA amplicon sequencing. – PLoS One 15: e0227434.
Ritter, C. D. et al. 2019a. The pitfalls of biodiversity proxies: Differences in richness patterns of birds, trees and understudied diversity across Amazonia. – Sci. Rep. 9: 1–13.
Ritter, C. D. et al. 2019b. Locality or habitat? Exploring predictors of biodiversity in Amazonia. – Ecography (Cop.). 42: 321–333.
Ritter, C. D. et al. 2019c. Biodiversity assessments in the 21st century: The potential of insect traps to complement environmental samples for estimating eukaryotic and prokaryotic diversity using high-throughput DNA metabarcoding. – Genome in press.
Ritter, C. D. et al. 2020. Advancing biodiversity assessments with environmental DNA : Long-read technologies help reveal the drivers of Amazonian fungal diversity. 00: 1–16.
Tedersoo, L. et al. 2021. The Global Soil Mycobiome consortium dataset for boosting fungal diversity research. – Fungal Divers. 111: 573–588.
Ward, B. B. 2002. How many species of prokaryotes are there? – Proc. Natl. Acad. Sci. 99: 10234–10236.
Wearn, O. R. et al. 2012. Extinction debt and windows of conservation opportunity in the Brazilian Amazon. – Science (80-. ). 337: 228–232.
Yilmaz, P. et al. 2014. The SILVA and “all-species Living Tree Project (LTP)” taxonomic frameworks. – Nucleic Acids Res. 42: 643–648.
 Camarões Marinhos Reprodução, Maturação e Larvicultura (Livro Impresso)
R$50,00
Camarões Marinhos Reprodução, Maturação e Larvicultura (Livro Impresso)
R$50,00
 Manual de Boas Práticas: Qualidade e Segurança para Bons Negócios (livro digital - PDF)
R$0,00
Manual de Boas Práticas: Qualidade e Segurança para Bons Negócios (livro digital - PDF)
R$0,00
 Recursos naturais na vida caiçara (livro digital - PDF)
R$0,00
Recursos naturais na vida caiçara (livro digital - PDF)
R$0,00
 A produção integrada na carcinicultura brasileira (Volume 2) - (Livro Digital - PDF)
R$0,00
A produção integrada na carcinicultura brasileira (Volume 2) - (Livro Digital - PDF)
R$0,00
 Atlas Anatômico e Histológico do Caranguejo-uçá (Livro Impresso)
Atlas Anatômico e Histológico do Caranguejo-uçá (Livro Impresso)
 Fichas Técnicas Ilustradas - Sebrae/Projeto AquiNordeste (Livro Digital - PDF)
R$0,00
Fichas Técnicas Ilustradas - Sebrae/Projeto AquiNordeste (Livro Digital - PDF)
R$0,00
 As lendas na educação - Estórias do Baixo Sul e do Recôncavo Baiano (livro digital - PDF)
R$0,00
As lendas na educação - Estórias do Baixo Sul e do Recôncavo Baiano (livro digital - PDF)
R$0,00
 Cartilha de Rastreabilidade: Ostras cultivadas - Projeto AquiNordeste (Livro Digital - PDF).
Cartilha de Rastreabilidade: Ostras cultivadas - Projeto AquiNordeste (Livro Digital - PDF).
 Água na indústria: uma análise da visão do setor industrial... (Versão Digital - PDF)
R$0,00
Água na indústria: uma análise da visão do setor industrial... (Versão Digital - PDF)
R$0,00
 As lendas na educação caiçara (livro digital - PDF)
R$0,00
As lendas na educação caiçara (livro digital - PDF)
R$0,00
O GIA é um grupo de pesquisas científicas, tecnológicas e de inovação criado e sediado na Universidade Federal do Paraná. Sua principal missão é assumir um papel direto na interação entre setor acadêmico, a iniciativa privada e a sociedade civil em geral, transpondo os muros universitários, aplicando com excelência e eficiência, métodos técnico-científicos de produção de alimentos e na resolução de problemas ambientais, sociais e econômicos que dizem respeito aos mais diferentes setores da sociedade brasileira.
No dia 09 de fevereiro de 2021 foi finalmente assinado o Termo de Compartilhamento estabelecido entre a UFPR, através de...
O GIA acaba de lançar mais um livro. O tema da vez é o cultivo de peixes orgânicos Cada vez...
Copyright © Grupo Integrado de Aquicultura e Estudos Ambientais (GIA) | Desenvolvido por Abrigo Virtual
Copyright © Grupo Integrado de Aquicultura e Estudos Ambientais (GIA) | Desenvolvido por Abrigo Virtual